服务热线
178 0020 3020
在研究某个基因或者蛋白A的过程中,我们往往需要人为地使A蛋白在细胞中水平升高,从而 观察到其对细胞内其他蛋白或者基因的影响。那么我们如何使A蛋白水平升高呢?最简单的方法就是往细 胞中转染A蛋白的表达载体。下面科研狗将全面介绍如何构建一个蛋白载体,将有一些列的专题介绍。我 们以构建p53蛋白的表达载体为例,在本次专题中,介绍如何设计一个表达载体的引物。
1 得到p53的CDS序列
1.1 打开http://www.ncbi.nlm.nih.gov/ , 在下拉框里面 选择Gene数据库(图1.1),然后输入p53进行搜索。

图1.1
1.2 选择对应物种的p53基因,我们这里选择克隆人的p53基因(human), 可以根据需要,选择其他物种的p53(图1.2)。

图 1.2
1.3 在p53基因的页面中,有非常多的信息,我们往下找到mRNA and Proteins栏目。很多基因能够编码不同亚型的蛋白,存在多个isoform,因此要选择正确的isoform。我 们点击NM_xxxx的链接,NM代表mRNA,NP代表蛋白质。(图1.3)
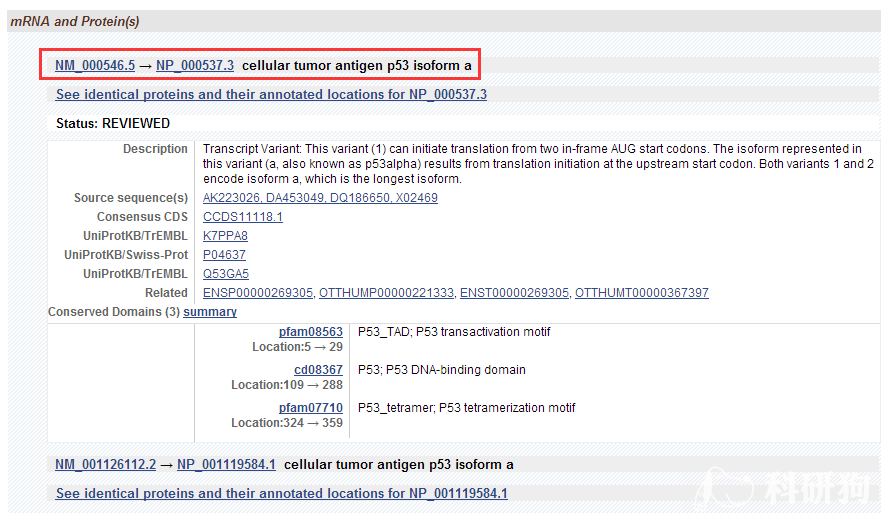
图1.3
1.4 在新的网页中,我们找到如下图的CDS链接并点击。(图1.4)

图 1.4
1.5 点击CDS链接之 后如下图所示。(图1.5)
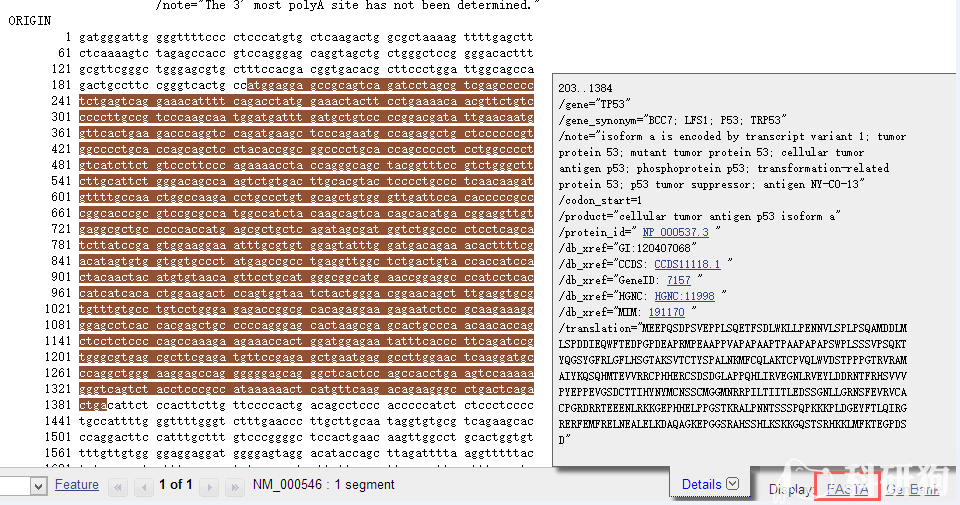
图1.5
1.6 接下来点击上图(图1.5)中的红色框“FASTA”,得到如下结果(图1.6) ,从ATG开始到TGA结尾为p53的CDS区。我们把这段序列粘贴到一个记事本中,用于后面操作。

图1.6
2 选择合适的表达载体
因为我们要表达蛋白,我们选择表达载体。比较常用的了PCI-neo, pCMV, pEGFP等,我们在 这选择PCI-neo。如何选择载体,我们将在后面的专题中介绍,或者可以登录科研狗网站 /upload。
3 确定双酶切位点
PCI-neo载体的多克隆酶切位点有:NheI, Xhol, EcoRI, Mlul, Xbal, Sall, Accl, Smal, Notl。 如图3.1红色框标记所示。
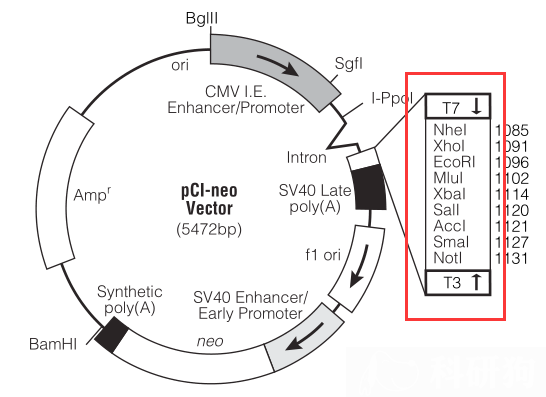
图3.1
选择双酶切的两个酶要满足如下条件:
(1) 两个酶在载体中存在。
(2) 两个酶不存在于目的蛋白的CDS中。
(3) 两个酶应该具有共同酶切的Buffer。
(4) 两个酶在载体上多克隆酶切位点位置最好不能太近。
如何满足上面条件呢?我们首先通过primer 5来确定,primer 5软件及教程可以在科研狗网站中找 到。
3.1 首先打开软件执行file > new > DNAsequence(图3.2)。
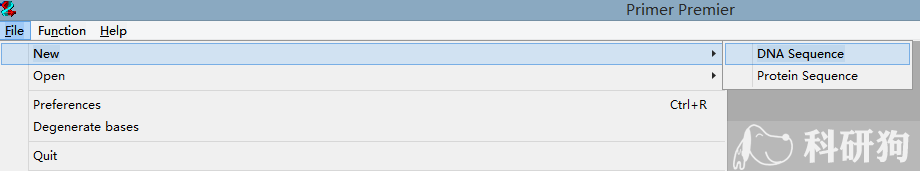
图3.2
3.2 把我们在步骤1.6中获得的p53的CDS序列粘贴在下图大的红色方框中,在弹 出的对话框中选择"As is",其他三个选项分别对应"反序","互补", "互补反序"。然后点击"Enzyme"图示中红色小框(图3.3)。
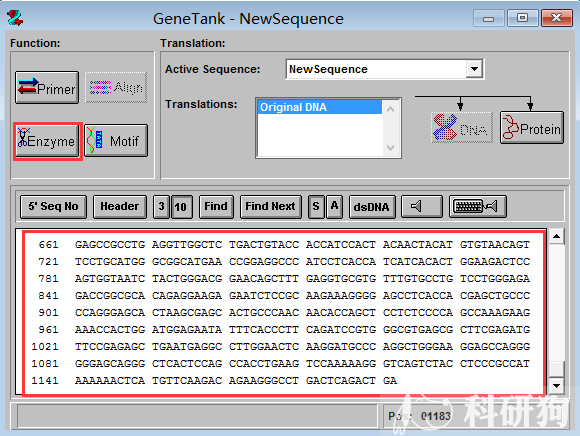
图3.3
3.3 在弹出的框中,首先把右边”selected enzymes“全部选中,点中间 的”del“删除掉,然后再在左边”All enzymes“里面把PCI-neo中的酶切位点选中,点击”ADD“添加 到右边。最终结果如图3.4所示。
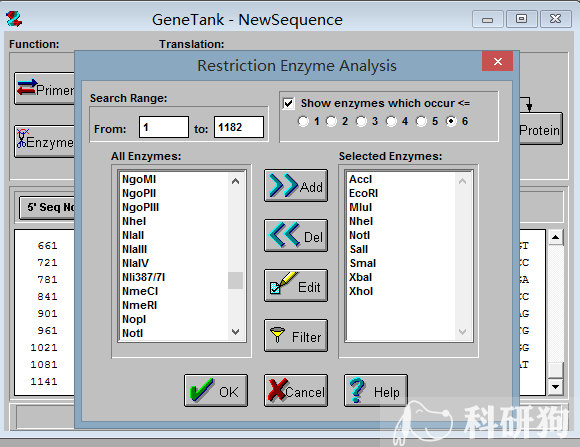
图3.4
3.4 然后点击OK,出现如下图,红色框标记的Accl 表示这个酶切位点存在于p53 序列中,因此除了Accl不能用,其他的暂时可用。(图3.5)
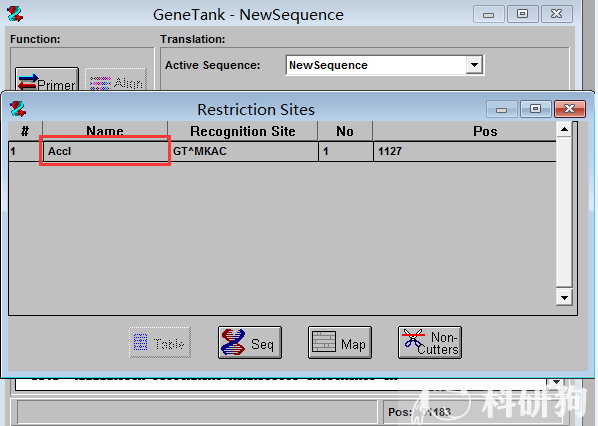
图3.5
3.5 因为我们采用的是双酶切,那么要求所用的两个酶都必须有相同的酶切BUFF ,这样才能在同一缓冲液中同时进行酶切。我们采用NEB的酶,可以打开http://www.neb-china.com/, 在页面最下面找到红色框(图3.6)。
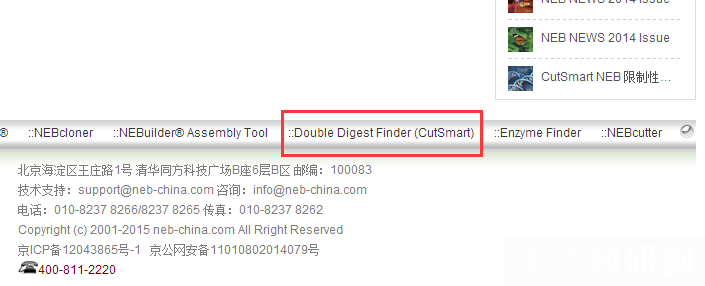
图3.6
3.6 在打开的页面中,我们把上面可用的酶两两进行查找(图3.7),如果你现 在拥有的是旧的BUFF(标记为1,2,3,4,新的系统为1.1, 2.1, 3.1)请点击图3.7中长红色框。找到具有 共同BUFF的两个酶,我们找到了XhoI和NotI。图3.8中表示NotI和 XhoI两个酶在BUFF 3.1中的活性均为100%,因此可以选择这两个酶。(在比较早的NEB产品中,BUFF均是 由1,2,3,4组成,如果你使用的是旧的BUFF,那么请采用旧的查找系统,新的BUFF已经加了BSA,因此可 以不用额外添加BSA)。

图3.7
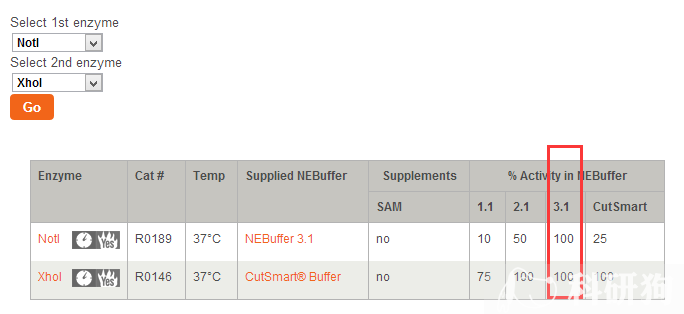
图3.8
3.7 我们确定了使用XhoI和NotI这两个酶切位点,那么我们找出这两个酶切位点的序列:
XhoI: C TCGA G
NotI: GC GGCC GC
我们最终要构建载体的示意图如下(图3.9)

图3.9
4. 获得p53的CDS区的PCR引物
我们计划从细胞中提取mRNA,然后逆转录成cDNA,然后从cDNA里面用引物把p53的CDS区扩增出来。
4.1 获得初步的上下游引物
表达载体和其他PCR引物有一点区别,因为我们要把p53的CDS区全部扩增出来,因此上下游引物的开 端是固定的。因此上游引物我们选取CDS区前20(最好25个)个碱基:
p53-上游引物:ATGGAGGAGCCGCAGTCAG
下游引物获得方法我们先 选择CDS区最后20(最好25个)个碱基的序列:GAAGGGCCTGACTCAGACTGA。然后把这段序列反向互补得到 新的序列:TCAGTCTGAGTCAGGCCCTTC。这个序列为下游引物序列。
4.2 判断获取的引物是否合适
上面得到的引物只是符合了匹配p53CDS区,在全细胞的从 DNA中进行PCR,我们还不知道他的特异性怎样。我们在NCBI数据库中检测这对引物的特异性。打开: http://blast.ncbi.nlm.nih.gov/Blast.cgi 选择下面红色框标识的Primer Blast(图4.1)
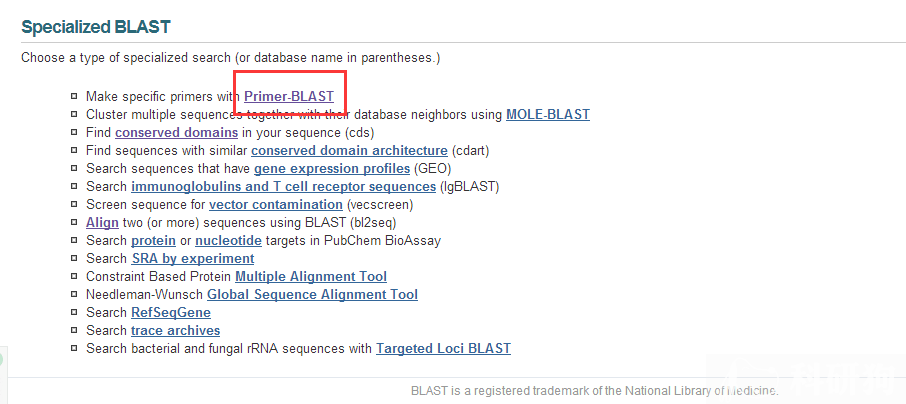
图4.1
4.3 在出来的网页中,把4.1中得到的两队引物分别输入,如图4.2。其他参数不要修改,然后 点击最下面的Get Primer.

图4.2
4.4 经过一段时间等待后,出来了如下页面(图4.3),该页面中100%匹配的只有p53,证明引 物特异性可以。如果引物的退火温度以及特异性不够,可以增加引物的长度。因此我们确定了p53的上下 游引物。

图4.3
5 把上游引物加上XhoI酶切位点,下游引物加 上NotI酶切位点
在构建表达载体的时候非常容易范的错误在于酶切位点添加不正确导致的移码突变,因此很多同学 测序发现100%正确,但是转染到细胞中就不能表达,这样的情况大部分是由于移码突变引起的。我们知 道蛋白质是由三个碱基连在一起形成的一个读码框决定的,所以一定要保证插入p53序列的读码框是从 ATG开始,如果一旦碱基A(或者AT)被前面的两个(一个)碱基组装在一起形成读码框,那么后面所有 的氨基酸都被改变了。
5.1 找到上游酶切位点(XhoI)在载体(PCI-neo)上的读码框分布情况。一般 载体上都会三个三个碱基标示好读码框,如下图所示(图5.1)。

图5.1
但是PCI-neo载体上面并没有三个碱基一起标示,如图5.2所示(图中红色的是我们后面添加上的) 。但是PCI-neo载体标记了一个T7 Transcription Start位点,图中红色箭头开始,我们可以三个三个碱 基把其标识出来。
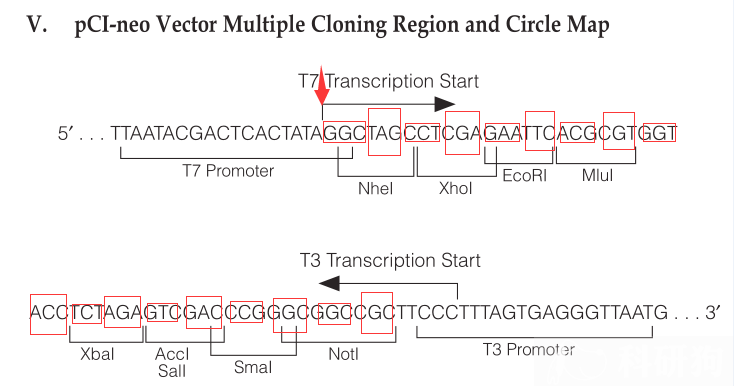
图5.2
5.2 假设我们直接把XhoI酶切位点序列加在p53上游引物中,将会有什么后 果呢,如下图所示(图5.3)。
XhoI中CGA(下图中红色框标识)为一个读码框,然后剩下的简介G会把p53的前面的AT给并过去,这 就发生移码突变了!
这种情况该如何解决呢?我们只需要在引物XhoI和p53上游引物间增加两个碱基,如下图中绿色的两 个碱基,这样一来保证了p53的ATG作为一个读码框。那么这两个碱基加什么好呢?本科研狗的观点是随 便加,只要不凑成一个终止密码子。
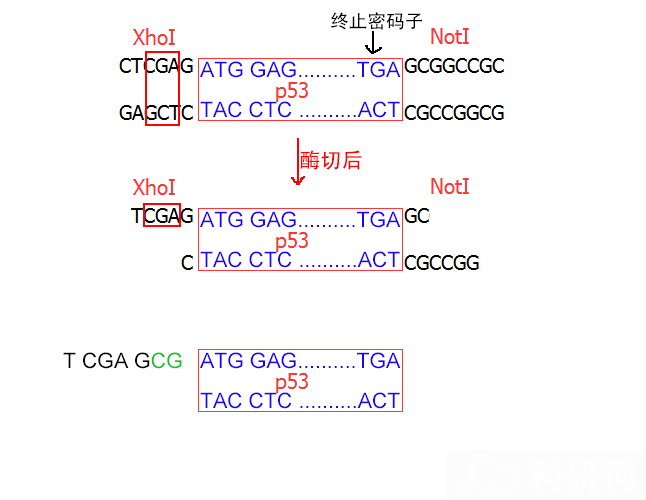
图5.3
5.3 我们得到了上游引物 CTCGAG CG ATGGAGGAGCCGCAGTCAG. 为了酶能够更好地识别PCR之后的酶切位点,我 们还必须在上面加上一段无义序列 比如ATA,GAG等(限制性内切酶更容易识别线性结构)。最终得到的 上游引物为:
p53-PCI-new-F: ATACTCGAG CG ATGGAGGAGCCGCAGTCAG
5.4 下游引物就比较简单了,直接在p53的下游引物前加上NotI酶切位点以及 保护碱基就可以。因为下游引物中存在终止密码子,因此无需考虑下游的移码突变,除非在下游有其他 的标签蛋白比如GFP,FLAG,HIS等标签蛋白,我们将在另外的专题中进行学习。
下游引物为:ATA GC GGCC GC TCAGTCTGAGTCAGGCCCTTC
至此我们已经得到了p53构建到PCI-neo载体中的引物~接下来就是PCR酶切连接的实验了,我们将继 续学习~有问题可以随联系我们
附件