服务热线
178 0020 3020
向表达载体中插入目的基因进行表达是个技术活~里面充满了各种需要注意的地方,
【案例】
使用双酶切的方式将一段基因序列插入pDsRed2-N1质粒中,使其能够与DsRED蛋白进行融合表达。
主要涉及到以下几个方面知识点:

1. 序列导入
pDsRed2-N1质粒可以直接在SnapGene的序列库中找到,导入方式在前面的文章中已经介绍,这里不再赘述。从图谱中选择多克隆位点区域MCS,点击,并转到序列页面:
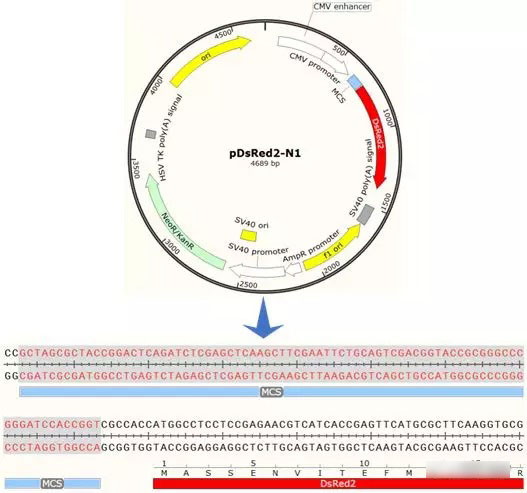
同时,新建DNA序列,将目的基因的序列粘贴进去,用于后续的酶切位点分析。
2. 酶切位点分析
双酶切连接的酶需要满足一下要求:
一、酶切位点需要存在于MCS中,但同时不能存在于目的基因序列中。
二、两个酶切位点之间至少要有几个碱基间隔,不能紧邻或重叠。
三、最好选择成熟的内切酶。
按照以上原则,我们先看基因中的常见酶切位点:
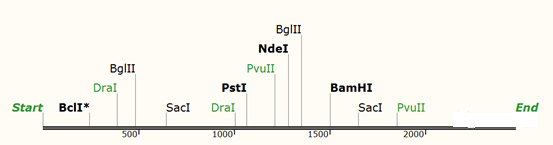
然后在MCS中排除这些酶切位点:
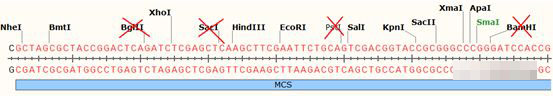
之后在剩下的酶切位点中,选择常用的即可,因为HindIII、EcoRI两个酶切位点靠的太近,小编没有选择这两个的组合,以免造成双酶切的效率下降。因此,小编最终选择的是EcoRI+KpnI的酶切组合。
3. 调整融合表达的阅读框
因为目的基因需要融合下游的荧光蛋白进行示踪,所以调整阅读框的通顺性是非常重要的,首先将目的基因中的终止密码子去除,然后将载体上EcoRI+KpnI之间的序列替换为去除终止密码子之后的目的基因的序列:
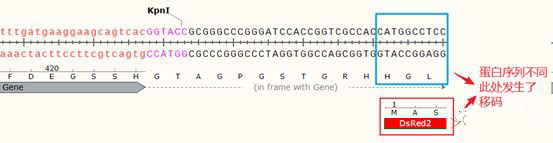
观察目的基因向下游表达至DsRed2中的氨基酸序列(HGL),发现与DsRed2原始序列(MAS)不同,即发生了移码,完整的阅读框遭到了破坏。因此,我们需要在目的基因下游与KpnI之间添加碱基恢复其阅读框的正确。

从上图中可以看出,当添加了两个碱基之后,之前的两个不同的氨基酸序列合二为一,变为正确的序列,即目的Gene可以跟下游的DsRed2基因进行正确的融合表达了。(如果您的蛋白序列3端不是核心功能区,去掉一个碱基同样可以修复阅读框)
4. 设计引物
有了上述方案,即可继续进行引物设计,进行扩增目的Gene全长了。
设计全长引物的软件有Primer5、Primer3Plus等,但小编更偏好于手动设计引物,在目的基因5端,用鼠标选中合适的区域(保护碱基-酶切位点-互补配对部分),当选择区域适合作为引物时,会实时显示出其Tm值:
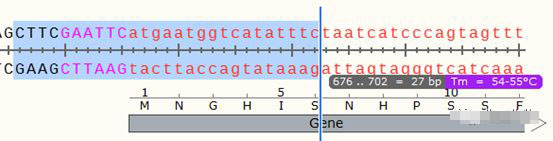
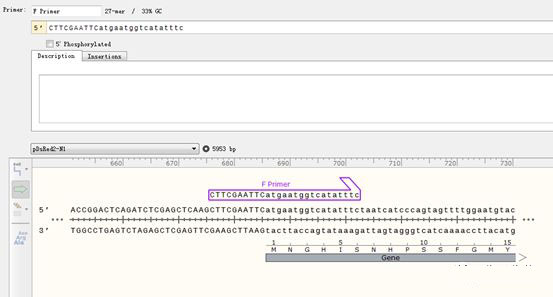
按Ctrl+R调出引物添加页面,正向引物选择Top Strand,正向引物就会显示在页面中。
使用相同的方法也可以设计反向引物(反向引物需要选择Bottom Strand):
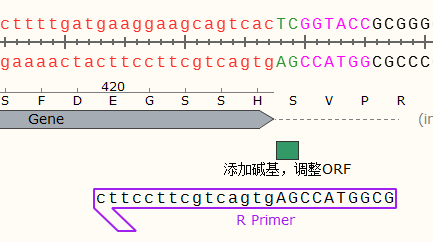
需要注意的是,扩增全长的引物5端通常情况下需要添加酶切位点和保护碱基,。而为了保证引物与模板的结合效率,互补配对部分又不宜少于15nt,因此整体引物会比较长,一般在25~35 nt,可以适当提升一下退火温度以提升扩增的特异性。
在图谱或者序列页面,按住Ctrl选中F Primer和R Primer,会将扩增产物的序列选中:
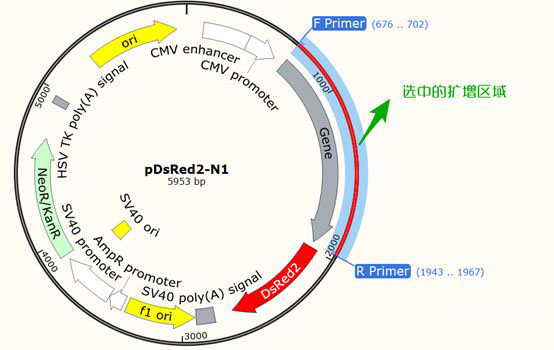
选中状态,Ctrl+C复制,然后在Word中Ctrl+V粘贴,就可以将引物连同产物一并整理到Word中了。

可见,使用合适的软件,可以使原本复杂繁琐的分析步骤变得简单,并且能够极大程度地提升效率。
附件