服务热线
178 0020 3020
随着CRISPR/Cas9技术的普及,单基因乃至单碱基的基因编辑已经逐渐成为实验室日常的技术体系。本文检验介绍如何使用最简单的方法对某一因进行基因敲除。
下面将以小鼠Mafb基因为例进行操作示范。
首先第一步时进行基因序列的获取,使用UCSC基因组浏览器找到Mafb的基因,
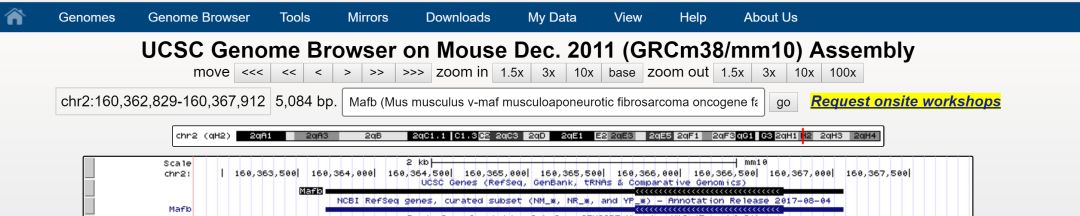
观察所需的基因信息,该基因只有一个外显子,并且位于负链的基因(转录方向为反向)
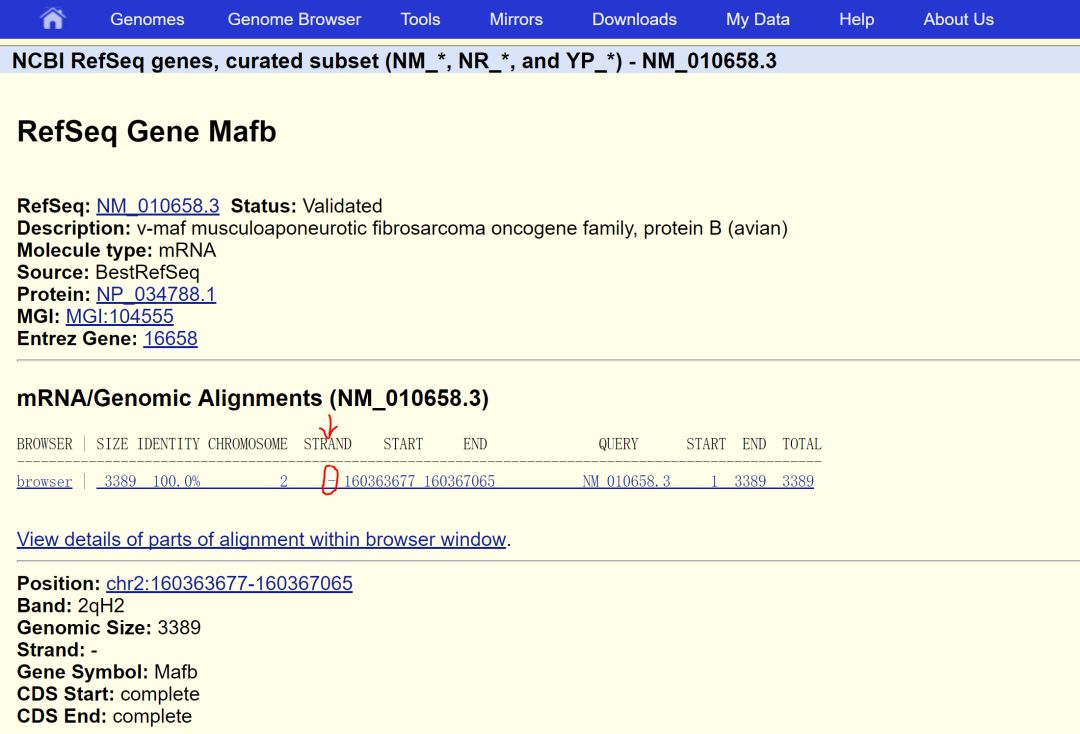
由于CIRSPR敲除机制的限制,在这里我们需要在含有CDS的第一个外显子的CDS区设计sgRNA,并且不能跨外显子。我们首先需要将基因信息复制到本地,建议保存到snapgene中进行后续操作。获取基因的方法使用UCSC的get gene
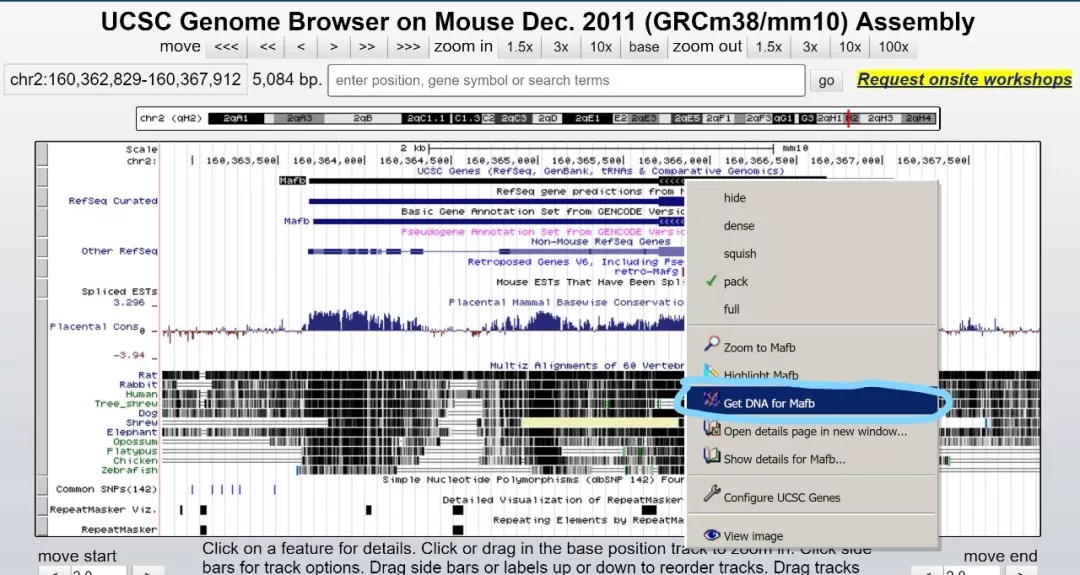
并且设置将基因上下游1000bp的区域一并获取。
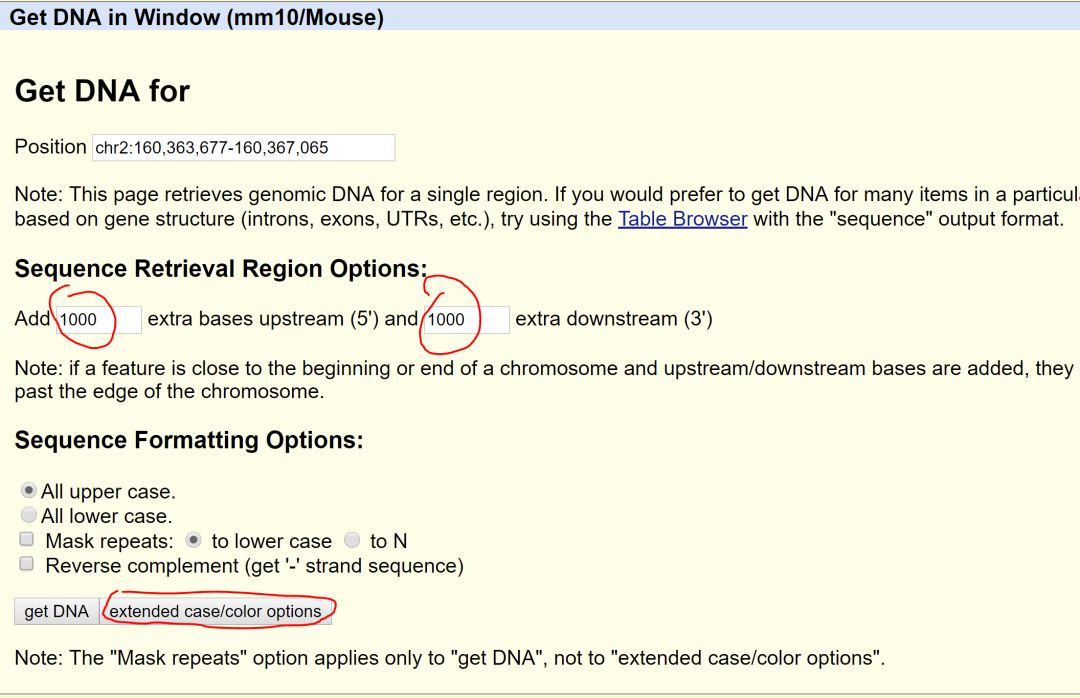
为了方便找寻基因区域,建议设置文字格式
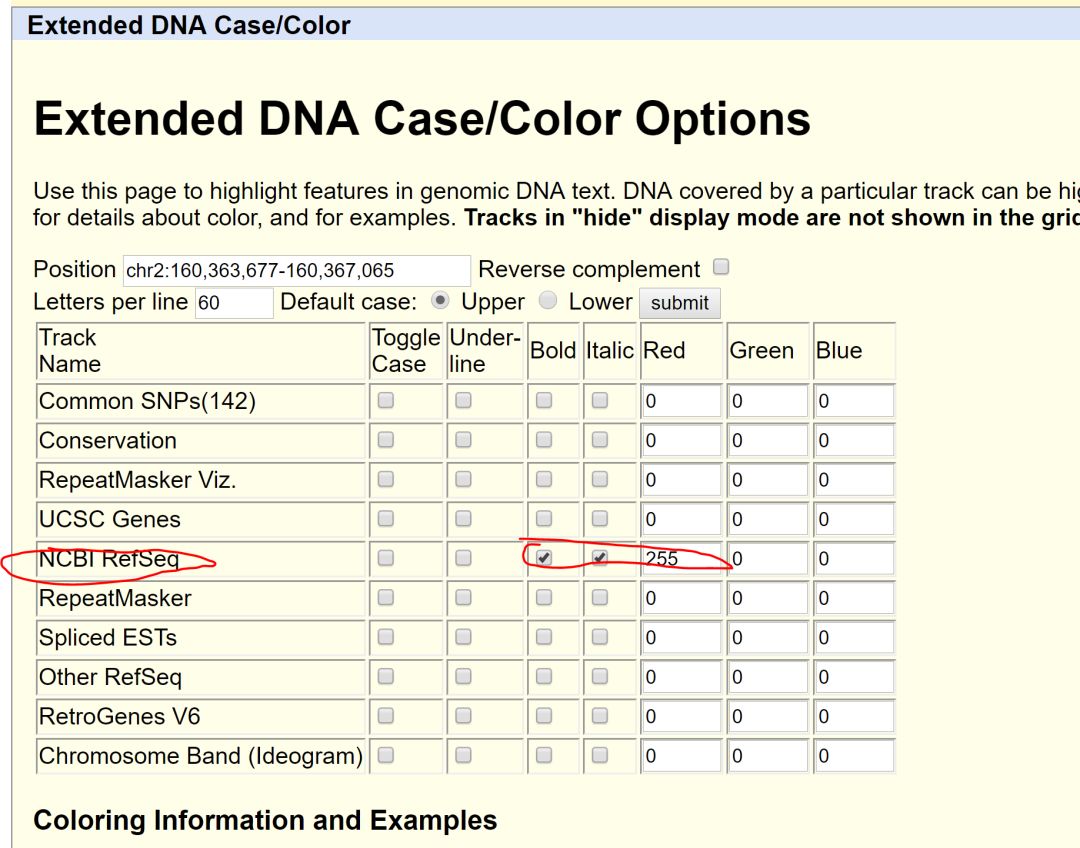
得到如下效果
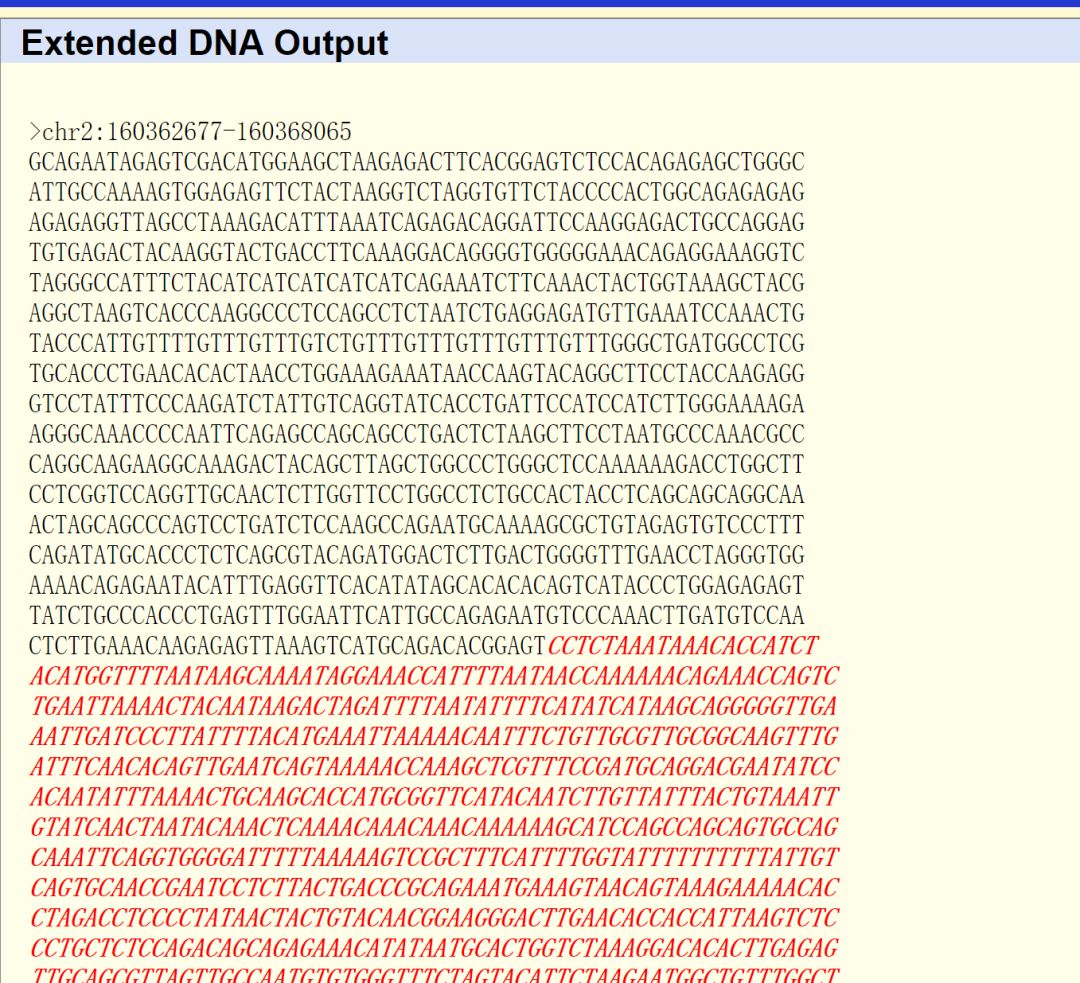
复制到snapgene中,并将序列进行翻转(view-flip sequence),将基因区域进行注释得到下面这样的注释信息
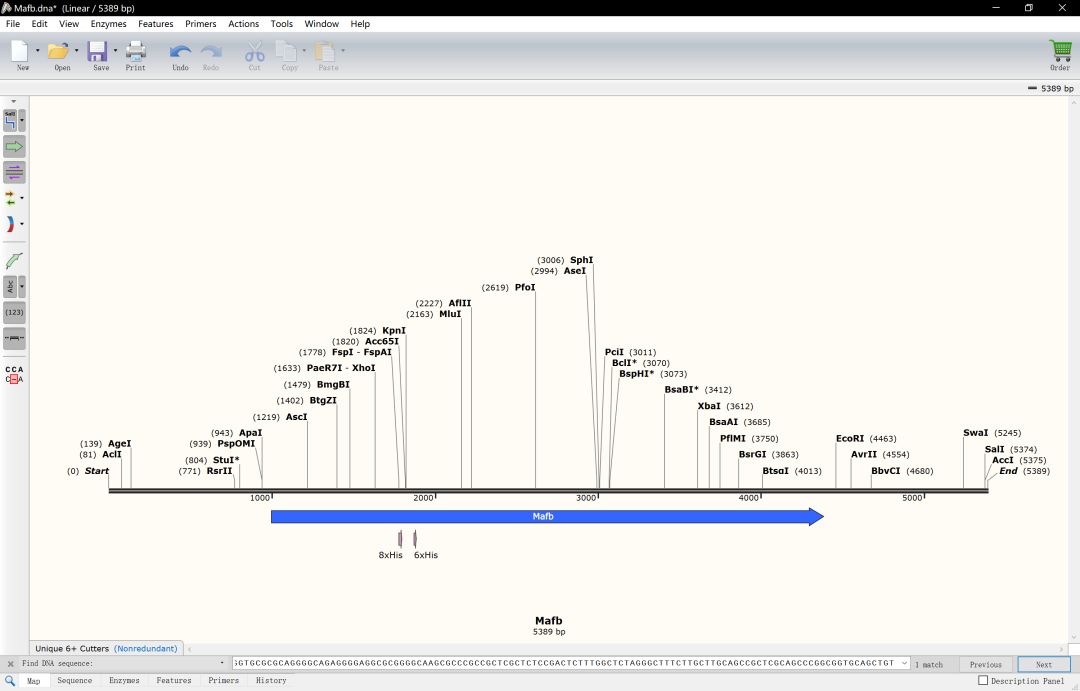
下一步对CDS区进行注释,使用NCBI 的基因组数据库找到Mafb基因
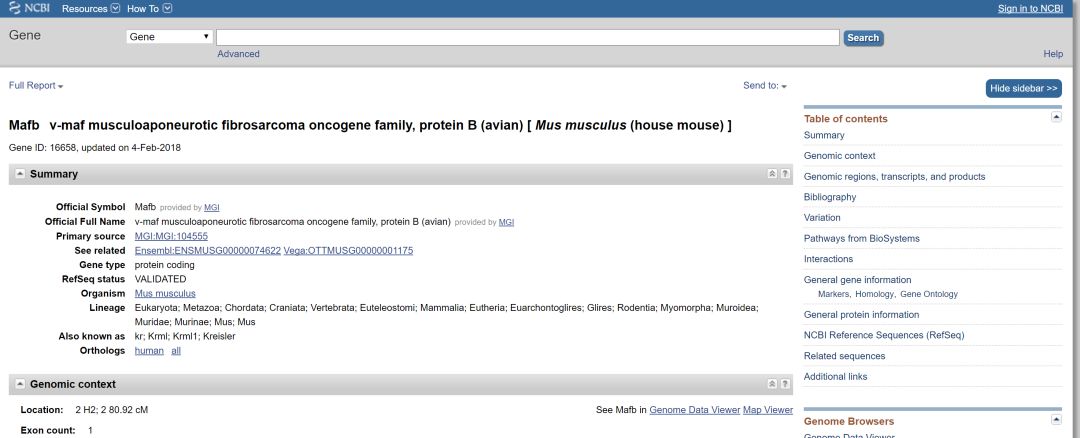
如果有多个蛋白亚型或者不同转录,都一并将信息手工添加到snapgene中
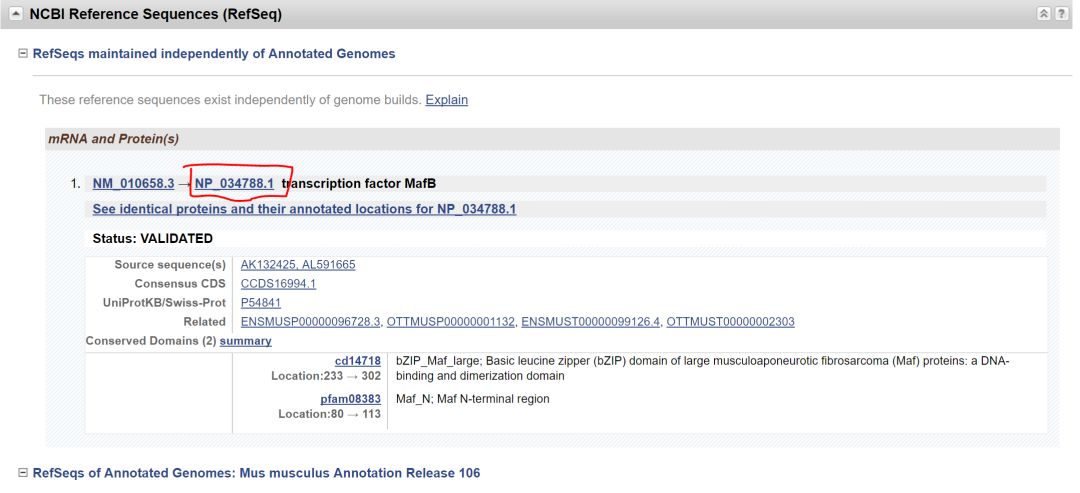
Mafb只有单一转录本和单一蛋白,下一步查找CDS区序列
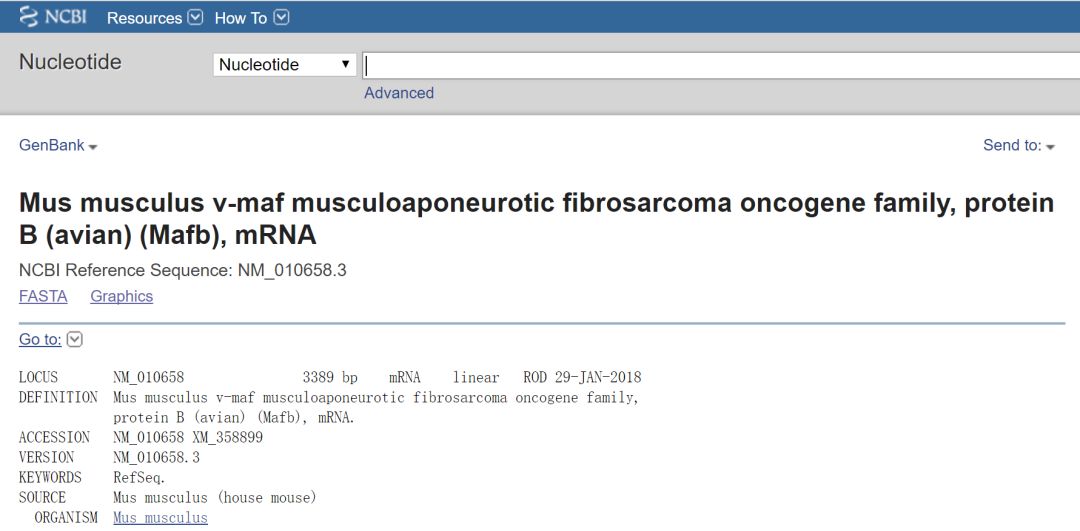
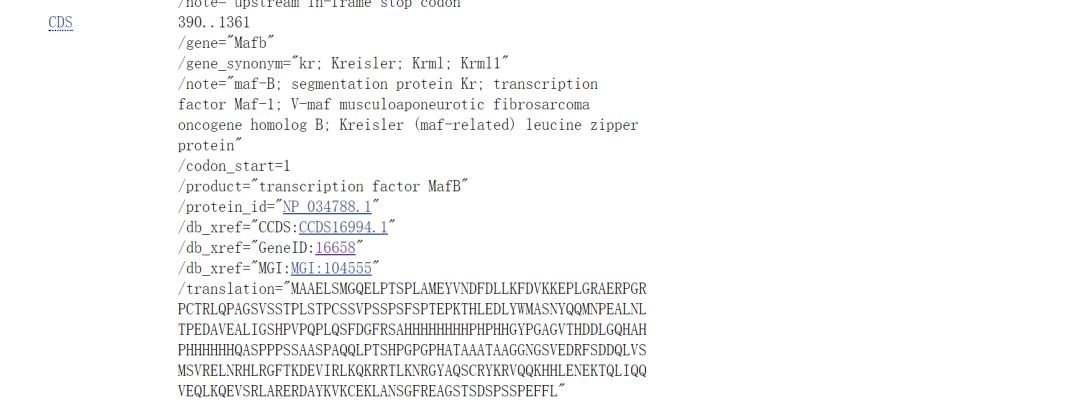
在下方根据CDS、起始位置选择CDS序列

在Snapgene中注释后如下
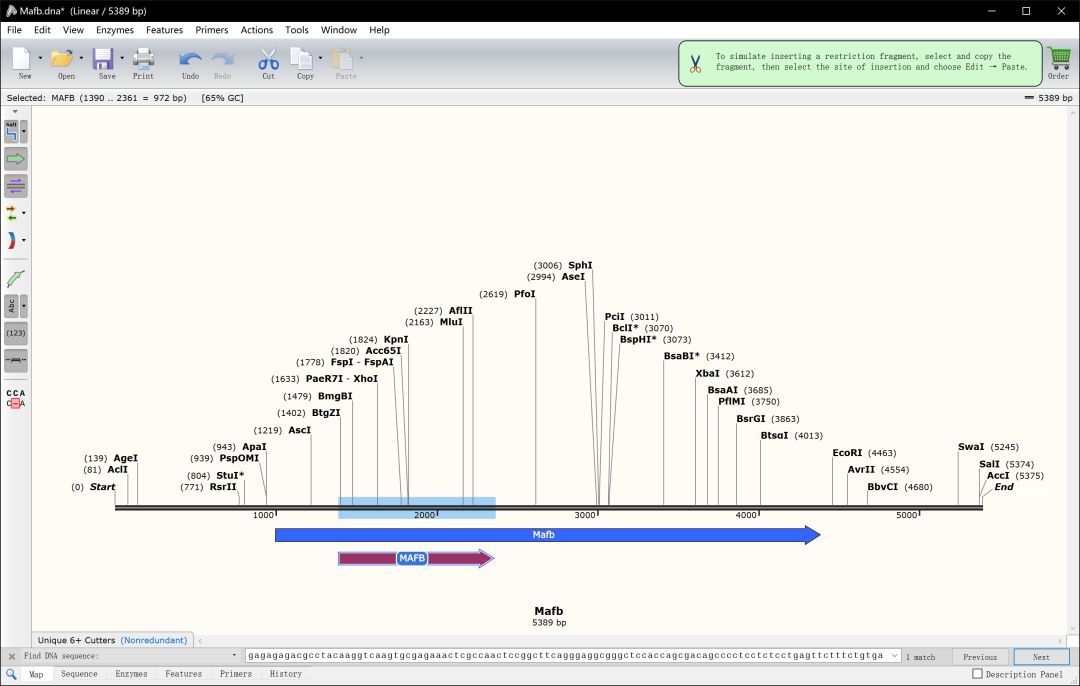
选择CDS前200bp的序列进行sgRNA设计,如果CDS前200bp序列跨外显子,则需要分开设计
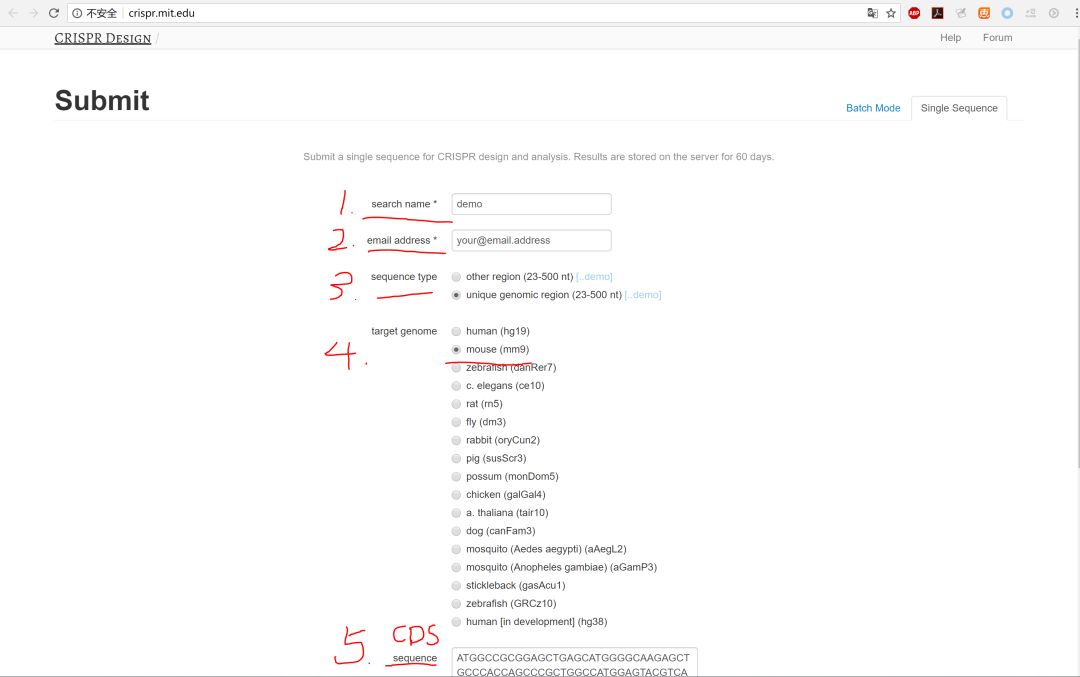
需要输入:1.name,2. email adress, 3. 序列类型,4.种属,5.序列
等待一会儿结果链接就会发送到邮箱
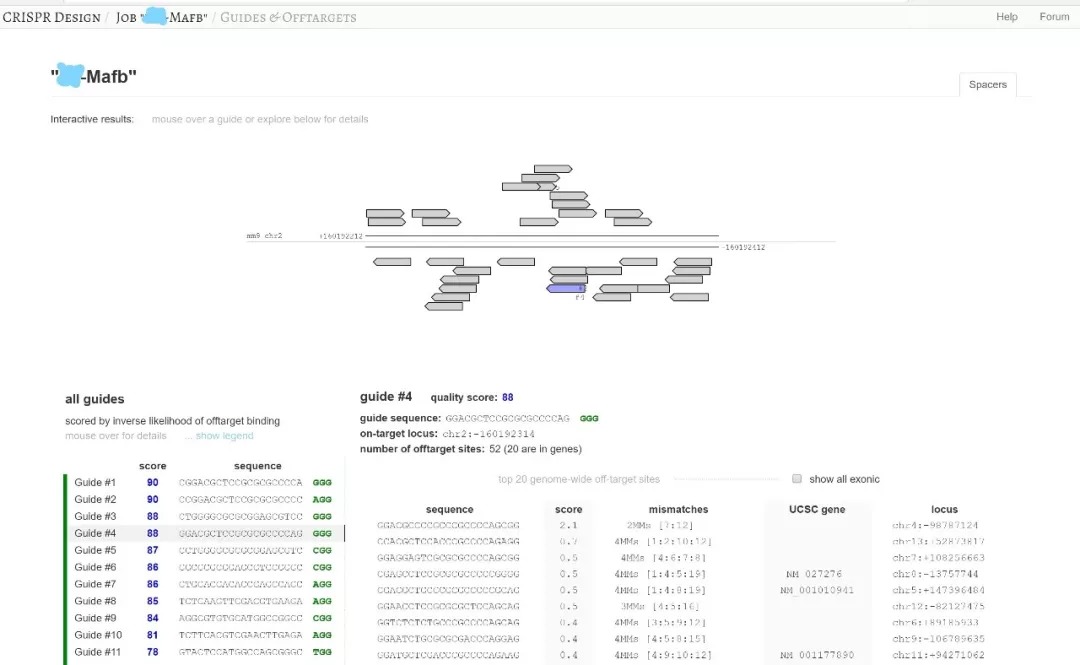
分析结果,选择最靠近5‘端及评分最高的sgRNA进行后续载体构建
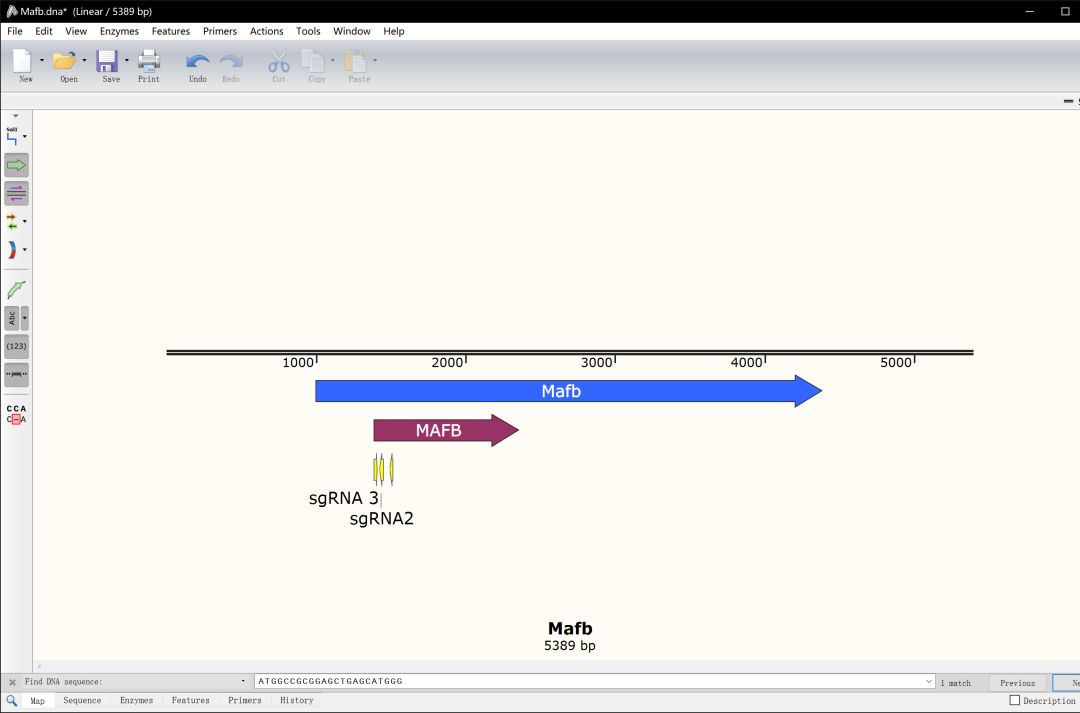
这里选择了#1,#11,#13进行构建,sgRNA位置如下
载体构建:
使用pGL3-U6-gRNA载体构建sgRNA表达载体,在Bsa1酶切位点将sgRNA序列进行插入
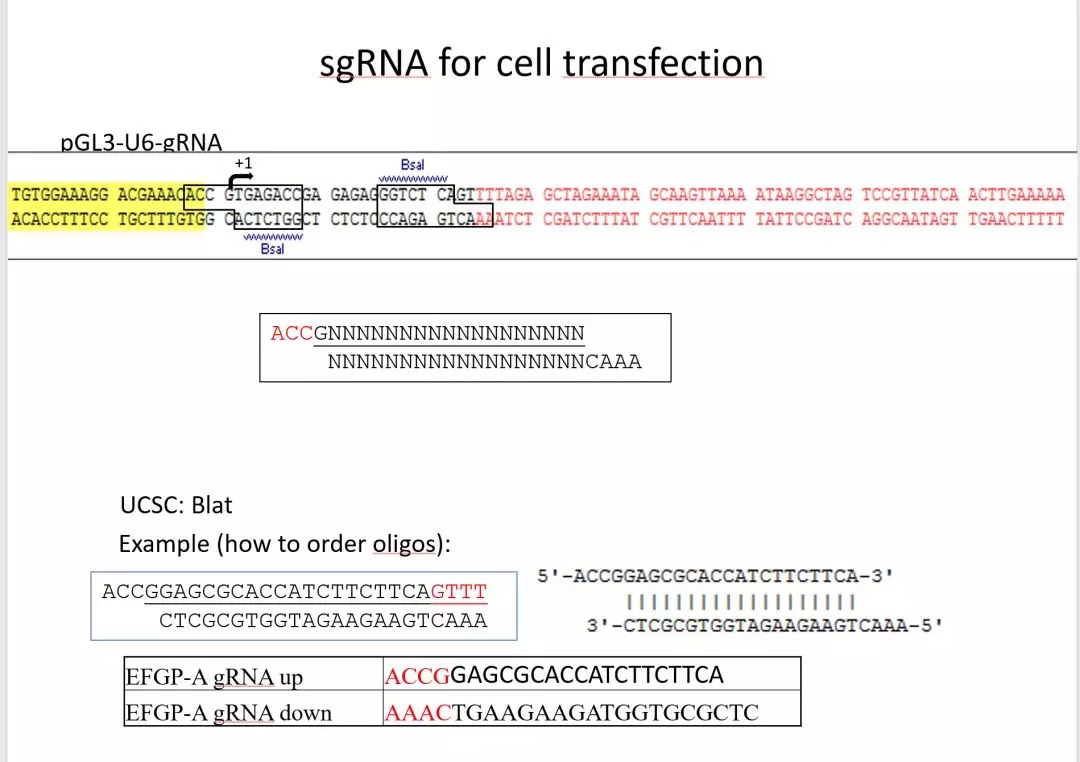
所需合成的oligo 片段如下

上下游oligo 退火后将形成粘末端,可直接(不需要纯化)与Bas1酶切过的载体(胶回收纯化)进行连接。测序成功后可以用于敲除。
其他载体包括单质粒系统的构建于此相似,不再详细阐述。个人感觉双质粒系统敲除效率更高,Cas9蛋白与sgRNA表达质粒的转染比例建议为1:3。
附件