IJBA | 上海交大陈海峰课题组发表核酸分子力场的最新研究成果
近日国际知名期刊International Journal of Biological Macromolecules在线发表了上海交大陈海峰教授课题组题为“Base-Specific RNA Force Field Improving the Dynamics Conformation of Nucleotide”的最新研究成果。该研究提出了一套碱基特异性的RNA分子力场(BSFF1),有效的提升了针对核酸的模拟效果。上海交通大学生命科学技术学院博士生李政新为该文章第一作者,生命科学技术学院本科毕业生穆俊羲为共同第一作者,陈海峰教授为通讯作者。

RNA作为中心法则的中心一环,在多个生理过程中都发挥了至关重要的作用,当前肆虐的新冠病毒也是一种RNA病毒。当前,人们对于RNA的研究远不如蛋白质的研究成熟,传统实验方法在解析RNA结构上也存在诸多困难,目前已解析的RNA结构数量(<6000)远小于蛋白质(>190000)。在此背景下,能够在原子层面研究生物大分子结构与动态变化的分子动力学模拟就成为了研究RNA的重要方法。然而,决定其精度的RNA分子力场仍存在较大不足,包括诸如过度稳定碱基堆积、zeta/alpha二面角表征失准、以及在模拟四核苷酸体系时容易产生错误的插层结构等问题。
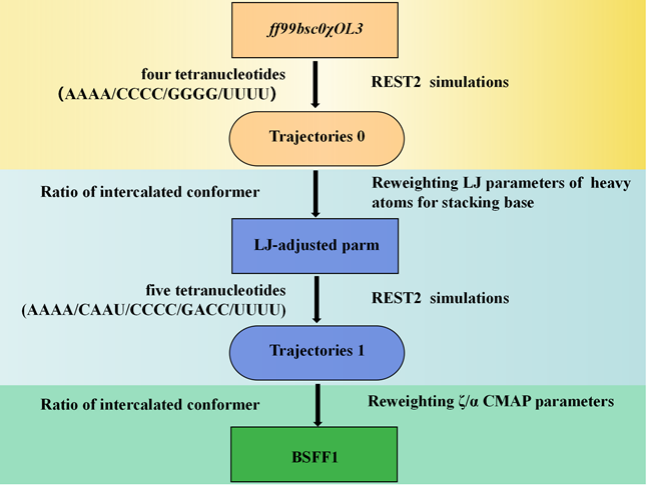
为解决上述不足,陈海峰团队通过重加权的方法(如上图),首先碱基特异性地修正了决定碱基堆积的非键参数,之后通过引入针对zeta/alpha二面角的格点能量矫正项(CMAP),极大的提升了对于四核苷酸体系的模拟效果。
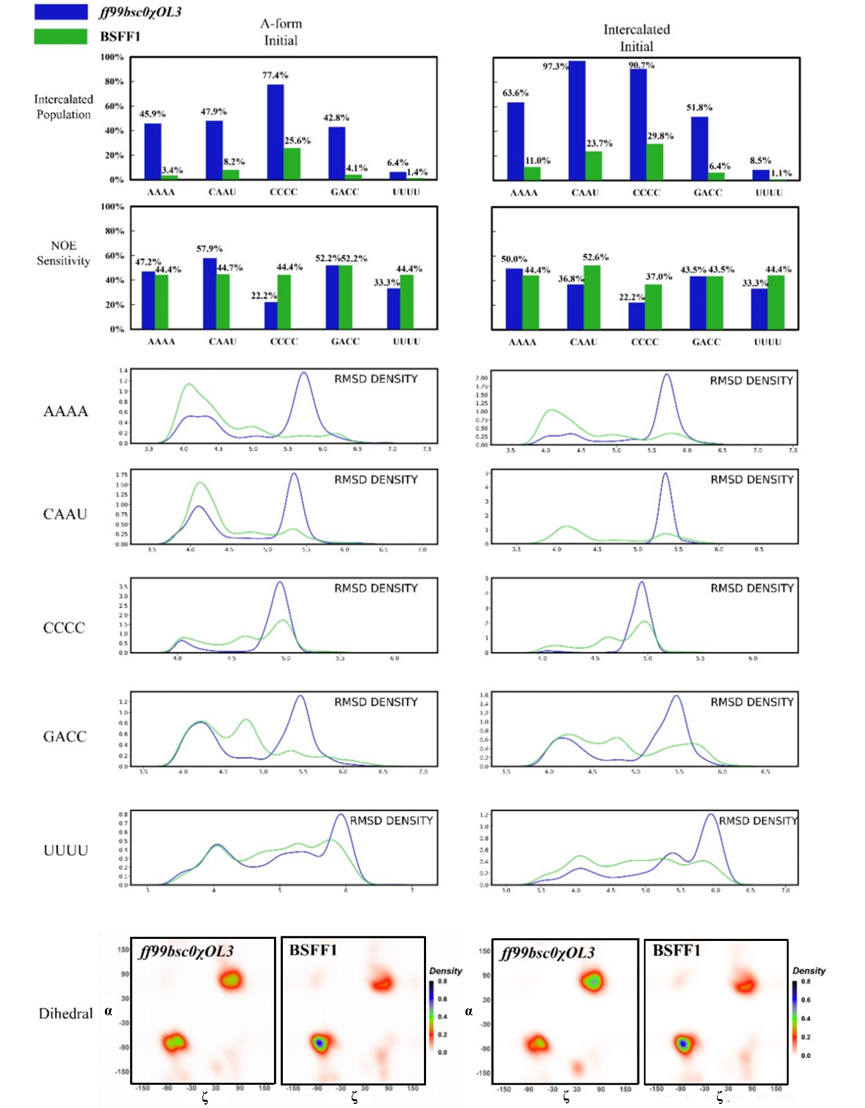
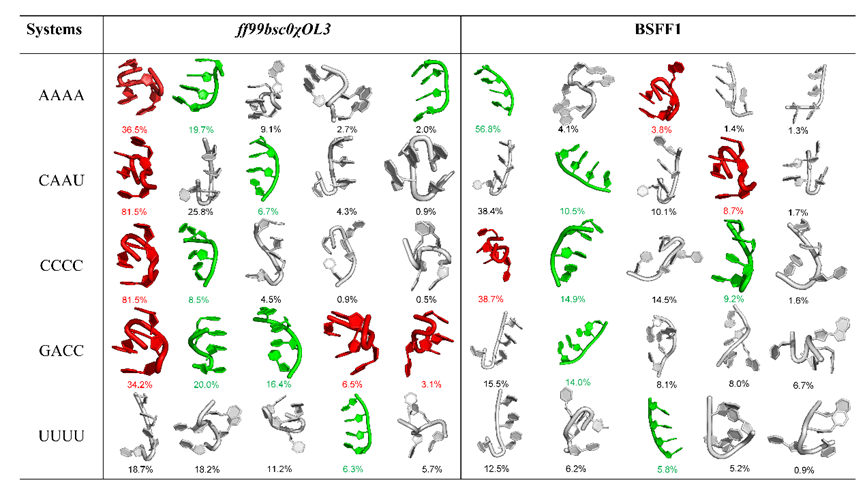
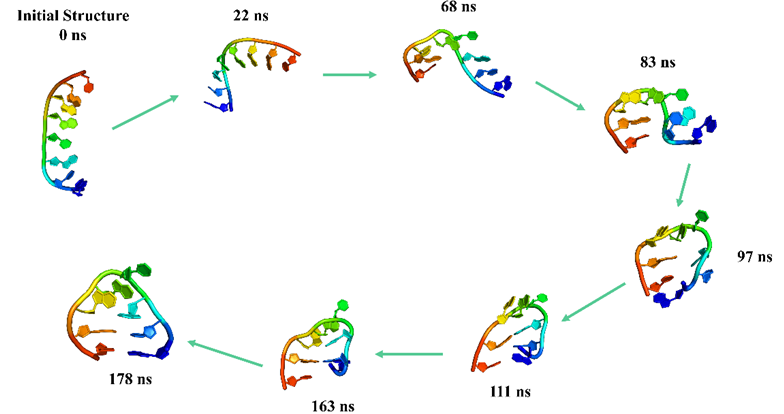
通过对于多个RNA常见体系如短单链、核糖开关、双螺旋、假结、k-turn等的模拟测试显示,该碱基特异性的RNA分子力场BSFF1能够稳定其实验结构。增强采样的结果显示,BSFF1能够将典型的RNA发夹(hairpin)进行从头折叠,将其从单链形式依序折叠为发夹形式,这进一步验证了BSFF1的全局合理性,同时对于研究RNA从头折叠以及核酸分子机制都有着先导提示作用。
该工作得到上海交通大学高性能计算中心(HPC)、国家自然科学基金项目(21977068和32171242)和中国国家重点研发计划(2020YFA0907700)的支持。
论文链接:
https://doi.org/10.1016/j.ijbiomac.2022.09.183