Genome Biology | 刘琦团队开发基于细胞类型空间邻域嵌入的多切片空间域智能解析平台
空间转录组测序技术的不断发展,对空间转录组数据的智能解析提出了重大挑战,其中对于空间域(Spatial domain)的识别尤为关键。空间域系指细胞在空间中形成的多细胞近邻结构(如肿瘤的三级淋巴结构,TLS),与特定的生物学功能密切相关。对空间域的深入解析对于理解生物体的发育过程、发现新的疾病标志物以及开发新的治疗方案等均有重要意义【1】。
2024年3月19日,同济大学刘琦教授课题组(博士后段斌,博士生陈绍奇、程小桔为文章共同第一作者)在Genome Biology线上发表了题为“Multi-slice Spatial Transcriptome Domain Analysis with SpaDo”的论文,发布了基于细胞类型空间邻域隐空间嵌入的多样本切片空间域智能解析平台SpaDo【2】。SpaDo同时适用于单细胞分辨率以及非单细胞分辨率的空间转录组数据,涵盖了针对空间域的三个功能分析模块:(1)单切片以及多切片空间域识别;(2)基于空间参考图谱的空间域注释;(3)基于识别的空间域进行多切片聚类分析。

目前主流的空间域解析方法主要关注于空间域的识别,可分为三类:(1) 不考虑空间信息的聚类算法,例如常用的Seurat和Scanpy等,这些方法通常被用作空间域识别性能评估的基准线;(2) 基于统计模型的方法,如BayesSpace,该方法基于“空间近邻细胞更有可能表现出相似的基因表达模式”这一假设进行贝叶斯推断,一般只适用非单细胞分辨率的空间转录组数据;(3) 基于图神经网络的方法,例如SpaGCN、SEDR、STAGATE等,该类方法基于细胞的基因表达信息,通过其近邻细胞重建的思想将细胞的基因表达信息和空间信息整合,获得每个细胞的表征。该类方法通常取得了较好的空间域识别效果。目前,上述研究工作大多仅适用于特定细胞分辨率或者单个切片的空间域识别。随着空间转录组数据的爆发式增长,迫切需要能够开发普适、有效的适合不同分辨率(单细胞分辨率以及非单细胞分辨率)、以及不同切片数量(单切片和多切片)的空间域智能解析的计算工具。
面向上述挑战,本研究工作开发了SpaDo,一个面向多切片空间域智能解析的计算平台。多细胞协作是实现复杂生物学功能的必要前提。研究者认为,具有相似功能的细胞应该在空间上具有某种相似模式的邻近分布,而单个细胞的功能则可由其所属的细胞类型来决定。因此,空间域的功能可以通过细胞类型在空间上的分布来进行刻画。基于这一朴素的思想,SpaDo提出了一种简单但有效的整合基因表达信息和空间信息的隐空间嵌入策略SPACE(SPatially Adjacent Cell type Embeddings),通过考虑细胞类型的邻域空间嵌入,来进行空间域的智能解析,包括:(1)单切片以及多切片空间域识别;(2)基于空间参考图谱的空间域注释;(3)基于识别的空间域进行多切片聚类分析。
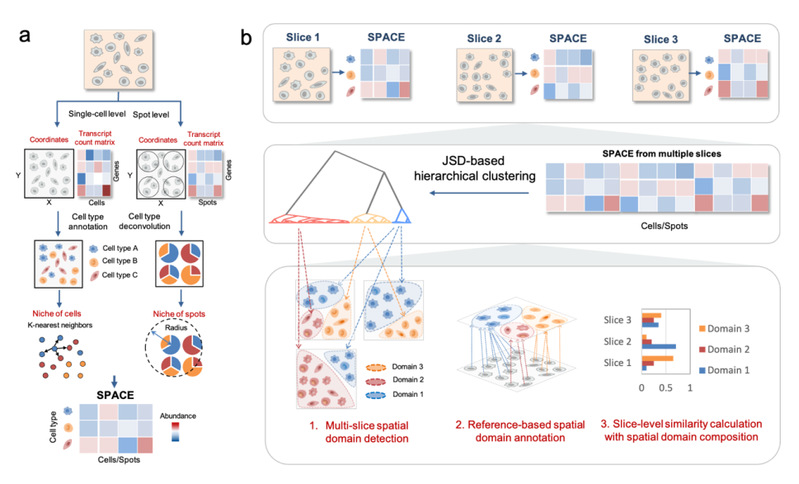
图1. SpaDo算法框架
相对于目前流行的基于图神经网络思想来整合基因表达信息和空间信息的方法,SpaDo提出的SPACE嵌入策略具有以下优势:(1)SPACE嵌入的特征单元是细胞类型,而细胞类型在多个切片之间能够一一对应,因此SPACE天然适用于对多个切片进行整合分析,无需额外的批次效应校正;(2)相较于复杂的图神经网络,SPACE基于空间近邻细胞类型组成,具有良好的可解释性,且计算复杂度较低,适合于大规模多切片数据分析;(3)SPACE具有更好的噪声鲁棒性。研究者发现,通过使用该简单朴素的嵌入策略,即可获得和复杂的图神经网络相当甚至更优的空间域识别效果,其可能的原因如下:(1)SPACE本质上采用了数据平滑策略,该策略虽然可能会弱化单个细胞空间信息,但对于整个空间域而言,其关键的空间信息,即空间近邻细胞类型组成,始终能够得到充分地保留;(2)平滑计算本身具有强大的抗噪能力,有利于在空间域识别中消除空间转录组数据中的噪声;(3)不同空间域的差别较为明显,无需精细尺度的表征即可识别。最近的两项研究均支持了研究者这一观点【3】【4】。
本研究进一步展示了利用SpaDo进行肿瘤三级淋巴结(TLS)的识别能力。同时,除了具备空间域识别这一功能之外,SpaDo可以基于空间参考图谱进行空间域注释(Spatial assignment), 以及进行多切片聚类分析(如对多个肿瘤切片进行聚类和分型等),进一步拓展了空间组学的解析模式和应用场景。
刘琦教授课题组长期从事AI驱动的组学智能解析和精准干预研究。本次工作是刘琦教授课题组在空间组学解析领域的又一有益探索。
参考文献
【1】Zeng Z, et al. Statistical and machine learning methods for spatially resolved transcriptomics data analysis. Genome Biol 23, 83 (2022).
【2 】Duan B, et al. Multi-slice spatial transcriptome domain analysis with SpaDo. Genome Biol 25, 73 (2024).
【3】Lin S, et al. Spatially resolved gene expression is not necessary for identifying spatial domains. bioRxiv. 10.15.562443 (2023). https:// doi. org/ 10. 1101/ 2023. 10. 15. 562443.
【4 】Singhal V, et al. BANKSY unifies cell typing and tissue domain segmentation for scalable spatial omics data analysis. Nat Genet. 56(3):431–41 (2024).