单细胞转录组学在单个细胞水平上对转录组进行测序分析,对于理解细胞和组织的复杂机制不可或缺。近年来,随着单细胞测序技术的快速发展,单细胞测序技术实验通量大幅增加,可以对愈加复杂多样的样本进行分析,并随着时间的推移积累了大量的参考数据集。各种社区甚至产生了许多图谱级单细胞数据集,如人类细胞图谱[1],人类肿瘤图谱网络等[2]。这些数据有助于以有监督训练模型的方式自动进行细胞类型鉴定,而无需预先标记基因注释。整合多源多机构的单细胞参考数据集有助于提高细胞类型鉴定效果,然而,不同机构或国家之间的组学数据传输和共享所涉及的隐私和政治问题正逐渐引起人们的关注。世界各国都在加强法律,通过禁止某些数据跨国家或跨组织传输来保护数据隐私和安全。这些法规包括欧盟实施的《通用数据保护条例》(GDPR)[3],美国颁布的《健康保险流通与责任法案》(HIPPA)[4]等。
日前,同济大学生命科学与技术学院生物信息系刘琦教授团队和微众银行杨强教授,范立欣博士等合作在SCIENCE CHINA Life Sciences线上发表了题为“Privacy-preserving integration of multiple institutional data for single-cell type identification with scPrivacy”的论文,发布了基于联邦深度度量学习的单细胞参考图谱构建和单细胞细胞类型自动鉴定框架scPrivacy,能够以数据隐私保护的方式整合多机构数据集来构建大规模单细胞参考图谱,并进行单细胞细胞类型自动鉴定。

整合多机构数据集进行单细胞细胞类型自动鉴定需要解决数据隐私保护问题,在不访问机构的原始数据的情况下整合多个机构数据集来训练泛化性能更好的模型,尽管目前已有一些整合数据集进行细胞类型鉴定的方法,所有这些方法都需要直接访问相关参考数据集,无法解决数据共享的相关法律限制和数据隐私保护问题,因而可能由于人类单细胞组学数据所存在的隐私和安全问题而不可用。
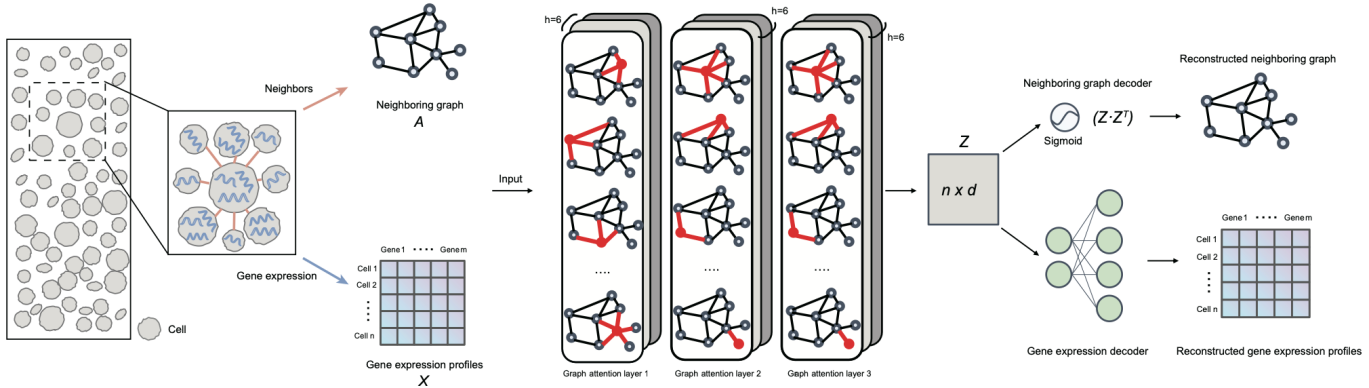
为解决单细胞数据整合时遇到的隐私保护问题,作者团队提出了一种联邦学习范式下的深度度量学习的单细胞细胞类型自动鉴定框架scPrivacy,以数据隐私保护的整合方式构建单细胞参考图谱,并进行单细胞细胞类型识别。联邦学习是隐私保护计算社区中的一种协作范式,它通过让用户在本地训练模型,服务器聚合各个用户的模型参数后训练全局模型,并将更新后的全局模型发送给各个用户,多次迭代直至收敛后最终生成稳定的全局模型,从而达到数据不出本地,只需要使用模型参数而不需要直接使用用户数据的效果[5]。scPrivacy的核心思想是通过联邦学习让每个机构在本地训练自己的模型并为所有机构聚合加密模型参数,以避免需要获取所有机构的单细胞原始数据,从而解决数据隐私保护问题,同时通过深度度量学习为每个单细胞数据集学习合适的度量,解决单细胞数据集异质性的问题(图一)。
该工作通过在27个基准数据集和15个来自不同医院的新冠病人的单细胞数据集上进行基准测试[6],证明了整合多机构数据集构建大规模单细胞参考图谱的必要性以及基于联邦深度度量学习的scPrivacy框架整合单细胞数据集的有效性。同时,scPrivacy由于其分布式训练的特性在时间消耗方面非常高效,并随着机构数据集数量的增加,其单细胞类型自动识别精度越来越高,同时对于不同的相似性度量、数据异质性和数据量大小具有鲁棒性。总结来说,该工作为隐私保护下构建大规模单细胞图谱提供了首个计算方法学和理论框架层面的参考和借鉴。

图Privacy的算法框架
刘琦教授课题组长期从事基于人工智能和组学数据挖掘的复杂疾病精准医学研究工作。近年来同时关注生物组学数据分析的隐私计算。课题组于2020年发表了药物定量构效关系建模领域的首个隐私保护计算框架FL-QSAR[7], 本次工作是课题组在单细胞组学隐私计算领域的又一有益探索。该论文第一作者是刘琦教授课题组的博士生陈绍奇和段斌,通讯作者是刘琦教授和微众银行的杨强教授,范力欣博士。该研究成果得到国家科技部重点研发计划BT&IT专项以及微众学者计划的资助。
1.Regev, A., et al., The Human Cell Atlas. eLife, 2017. 6.
2.Rozenblatt-Rosen, O., et al., The Human Tumor Atlas Network: Charting Tumor Transitions across Space and Time at Single-Cell Resolution. Cell, 2020. 181(2): p. 236-249.
3.Politou, E., E. Alepis, and C. Patsakis, Forgetting personal data and revoking consent under the GDPR: Challenges and proposed solutions. Journal of Cybersecurity, 2018. 4(1).
4.Benefield, H., G. Ashkanazi, and R.H. Rozensky, Communication and records: Hippa issues when working in health care settings. Professional Psychology: Research and Practice, 2006. 37(3): p. 273-277.
5.Yang, Q., et al., Federated machine learning: Concept and applications. ACM Transactions on Intelligent Systems and Technology (TIST), 2019. 10(2): p. 1-19.
6. Ren, X. et al. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas. Cell 184, 1895-1913 e1819 (2021).
7. Shaoqi Chen., et al, FL-QSAR: a federated learning-based QSAR prototype for collaborative drug discovery, 36, Bioinformatics, 2020, 5492–5498.